L’antibiogramme est un outil fondamental en microbiologie clinique, permettant d’optimiser le traitement des infections bactériennes. Cette technique, développée il y a plusieurs décennies, reste aujourd’hui incontournable pour guider le choix des antibiotiques et lutter contre l’antibiorésistance. En déterminant la sensibilité des bactéries aux différents antibiotiques, l’antibiogramme permet d’adapter précisément le traitement à chaque patient. Son importance ne cesse de croître face à l’émergence de bactéries multirésistantes, véritable enjeu de santé publique. Découvrons les principes, les techniques et les applications cliniques de cet examen essentiel.
Principes fondamentaux de l’antibiogramme
L’antibiogramme repose sur un principe simple : évaluer la capacité d’un antibiotique à inhiber la croissance d’une bactérie in vitro . Pour cela, on met en contact la souche bactérienne isolée chez le patient avec différentes concentrations d’antibiotiques. L’objectif est de déterminer la concentration minimale inhibitrice (CMI), c’est-à-dire la plus faible concentration d’antibiotique capable d’empêcher la croissance visible de la bactérie.
Cette approche permet de classer les bactéries en trois catégories pour chaque antibiotique testé : sensible (S), intermédiaire (I) ou résistant (R). Une bactérie sensible sera inhibée par les doses habituelles de l’antibiotique, tandis qu’une bactérie résistante ne le sera pas, même à forte dose. La catégorie intermédiaire correspond à une sensibilité diminuée, nécessitant parfois une adaptation posologique.
L’antibiogramme s’inscrit dans une démarche globale de diagnostic microbiologique. Il est réalisé après l’identification de la bactérie responsable de l’infection, généralement par des techniques de culture et d’identification biochimique ou par spectrométrie de masse. Cette approche séquentielle permet d’orienter le choix des antibiotiques à tester en fonction de l’espèce bactérienne isolée.
L’antibiogramme est un outil indispensable pour optimiser l’antibiothérapie et lutter contre l’émergence de résistances. Il permet d’éviter l’utilisation d’antibiotiques à large spectre lorsqu’un traitement plus ciblé est possible.
Techniques de réalisation de l’antibiogramme
Plusieurs méthodes sont utilisées en routine pour réaliser un antibiogramme. Chacune présente des avantages et des inconvénients en termes de rapidité, de coût et de précision. Le choix de la technique dépend souvent du contexte clinique et des moyens du laboratoire.
Méthode de diffusion en gélose (Kirby-Bauer)
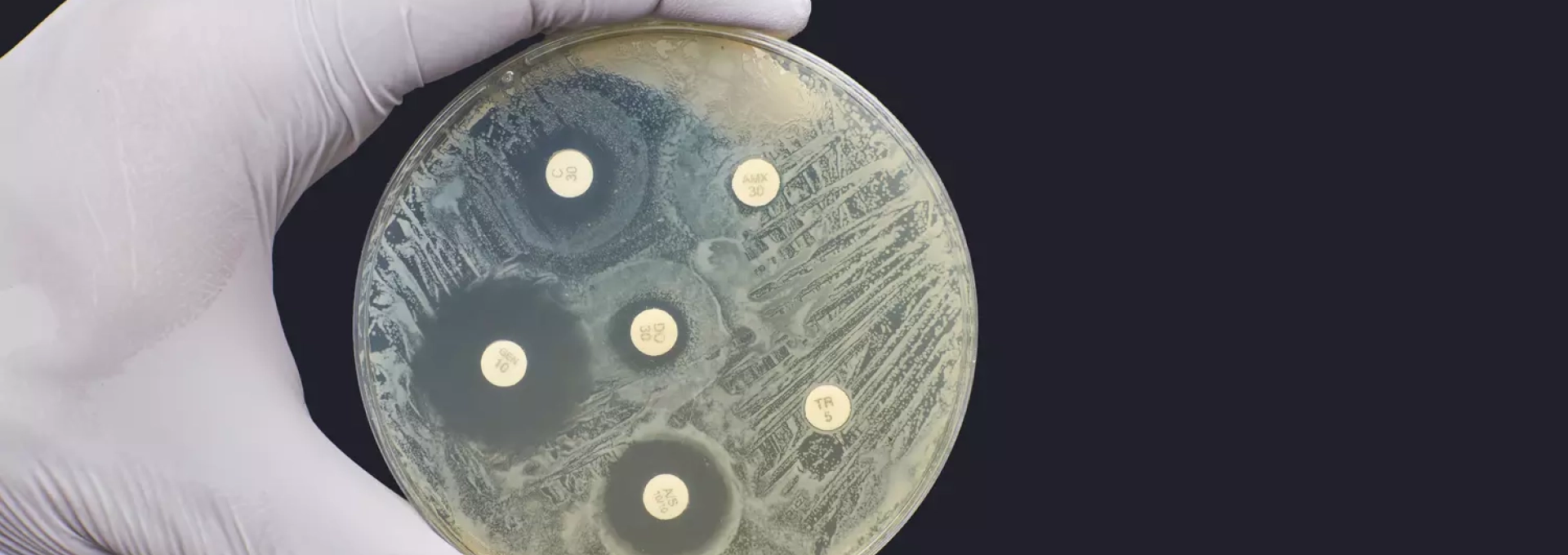
La méthode de diffusion en gélose, également appelée méthode des disques ou technique de Kirby-Bauer, est la plus ancienne et reste très utilisée. Elle consiste à déposer des disques imprégnés d’antibiotiques sur une gélose ensemencée avec la bactérie à tester. L’antibiotique diffuse dans la gélose, créant un gradient de concentration. Après incubation, on mesure le diamètre de la zone d’inhibition autour de chaque disque. Plus ce diamètre est grand, plus la bactérie est sensible à l’antibiotique.
Cette technique présente l’avantage d’être simple à mettre en œuvre et peu coûteuse. Elle permet de tester simultanément de nombreux antibiotiques. Cependant, elle ne fournit qu’une estimation semi-quantitative de la sensibilité et nécessite une standardisation rigoureuse des conditions expérimentales.
Méthode de dilution en milieu liquide (CMI)
La méthode de dilution en milieu liquide est considérée comme la technique de référence pour déterminer précisément la CMI. Elle consiste à mettre en contact la bactérie avec des concentrations croissantes d’antibiotique dans une série de tubes. Après incubation, on détermine la concentration minimale qui inhibe totalement la croissance bactérienne.
Cette méthode fournit des résultats quantitatifs précis, mais elle est plus laborieuse et coûteuse que la diffusion en gélose. Elle est principalement utilisée pour les bactéries difficiles à cultiver ou pour confirmer des résultats douteux obtenus par d’autres techniques.
E-test et bandelettes à gradient
Les E-tests et les bandelettes à gradient combinent les avantages de la diffusion en gélose et de la dilution en milieu liquide. Il s’agit de bandelettes imprégnées d’un gradient de concentration d’antibiotique. Appliquées sur une gélose ensemencée, elles permettent de lire directement la CMI à l’intersection entre la zone d’inhibition et la bandelette.
Cette technique est simple d’utilisation et fournit des résultats quantitatifs. Elle est particulièrement utile pour les bactéries à croissance lente ou exigeantes. Cependant, le coût des bandelettes limite son utilisation systématique.
Systèmes automatisés (VITEK, phoenix)
Les systèmes automatisés comme VITEK ou Phoenix permettent de réaliser simultanément l’identification bactérienne et l’antibiogramme. Ils utilisent des cartes ou des panneaux contenant des substrats biochimiques et des antibiotiques à différentes concentrations. La croissance bactérienne est suivie par des mesures optiques, permettant une lecture rapide et standardisée des résultats.
Ces systèmes offrent l’avantage d’une grande rapidité (résultats en 6 à 12 heures) et d’une standardisation poussée. Ils sont particulièrement adaptés aux laboratoires traitant un grand nombre d’échantillons. Leur principal inconvénient est le coût d’investissement initial élevé.
Interprétation des résultats de l’antibiogramme
L’interprétation des résultats de l’antibiogramme est une étape cruciale qui nécessite une expertise microbiologique. Elle permet de traduire les données brutes en informations cliniquement pertinentes pour guider le traitement.
Lecture des diamètres d’inhibition
Pour la méthode de diffusion en gélose, l’interprétation repose sur la mesure des diamètres d’inhibition. Ces diamètres sont comparés à des valeurs seuils définies pour chaque couple antibiotique/bactérie. Ces seuils, régulièrement actualisés, sont établis par des comités d’experts en tenant compte des données pharmacocinétiques et pharmacodynamiques des antibiotiques.
La lecture des diamètres doit être réalisée avec précision, en tenant compte d’éventuelles zones de croissance intra-inhibition ou de colonies mutantes résistantes. Une lecture automatisée par caméra peut améliorer la reproductibilité des mesures.
Détermination des CMI
Pour les méthodes quantitatives (dilution, E-test), l’interprétation se base sur la valeur de la CMI. Comme pour les diamètres d’inhibition, des concentrations critiques sont définies pour classer la bactérie en sensible, intermédiaire ou résistante. Ces seuils tiennent compte des concentrations d’antibiotiques atteignables in vivo aux doses thérapeutiques usuelles.
La détermination précise des CMI est particulièrement importante pour certains antibiotiques à index thérapeutique étroit, comme les aminosides ou la vancomycine. Elle permet d’optimiser la posologie pour atteindre les concentrations efficaces tout en minimisant le risque de toxicité.
Classification sensible, intermédiaire, résistant (SIR)
La classification SIR est le résultat final de l’antibiogramme, directement utilisable par le clinicien. Elle synthétise les données brutes (diamètres ou CMI) en tenant compte des aspects pharmacologiques et cliniques. La catégorie « sensible » indique que le traitement a de bonnes chances de succès aux doses habituelles. La catégorie « résistant » suggère un fort risque d’échec thérapeutique. La catégorie « intermédiaire » correspond à une zone d’incertitude, où le succès thérapeutique est possible avec des doses élevées ou si l’antibiotique se concentre au site de l’infection.
L’interprétation de l’antibiogramme ne doit pas se limiter à une simple lecture de la classification SIR. Elle doit tenir compte du contexte clinique, du site de l’infection et des propriétés pharmacocinétiques des antibiotiques.
Critères EUCAST vs CLSI
Deux principaux systèmes de standardisation coexistent au niveau international : l’EUCAST (European Committee on Antimicrobial Susceptibility Testing) en Europe et le CLSI (Clinical and Laboratory Standards Institute) aux États-Unis. Ces comités définissent les méthodologies, les critères d’interprétation et les contrôles de qualité pour l’antibiogramme.
Bien que globalement concordants, ces deux systèmes peuvent présenter des différences dans les valeurs seuils pour certains couples antibiotique/bactérie. Ces écarts reflètent parfois des différences dans les pratiques cliniques ou les épidémiologies locales. L’harmonisation internationale des critères d’interprétation est un enjeu important pour faciliter la comparaison des données de résistance entre pays.
Mécanismes de résistance détectés par l’antibiogramme
L’antibiogramme permet non seulement de guider le traitement, mais aussi de détecter et caractériser certains mécanismes de résistance aux antibiotiques. Cette information est précieuse pour l’épidémiologie et la compréhension de l’évolution des résistances bactériennes.
Production de β-lactamases (BLSE, carbapénémases)
La production de β-lactamases est un mécanisme majeur de résistance aux antibiotiques de la famille des β-lactamines (pénicillines, céphalosporines, carbapénèmes). L’antibiogramme permet de détecter la présence de β-lactamases à spectre étendu (BLSE) ou de carbapénémases grâce à des profils de résistance caractéristiques et à des tests complémentaires spécifiques.
Par exemple, la présence d’une BLSE se traduit typiquement par une résistance aux céphalosporines de 3e génération associée à une synergie entre ces molécules et l’acide clavulanique (inhibiteur de β-lactamases). La détection des carbapénémases repose sur l’observation d’une résistance aux carbapénèmes, confirmée par des tests phénotypiques ou moléculaires.
Résistance aux quinolones (QRDR, PMQR)
La résistance aux quinolones peut résulter de mutations chromosomiques dans les régions QRDR ( Quinolone Resistance-Determining Region ) ou de mécanismes plasmidiques (PMQR, Plasmid-Mediated Quinolone Resistance ). L’antibiogramme permet de détecter ces résistances par l’observation d’une diminution de sensibilité aux différentes quinolones testées.
Les mécanismes PMQR se traduisent généralement par une résistance de bas niveau, tandis que les mutations QRDR entraînent des résistances de haut niveau. L’interprétation fine du profil de résistance aux quinolones peut orienter vers le mécanisme impliqué et guider le choix thérapeutique.
Résistance inductible aux macrolides (phénotype MLSB)
La résistance inductible aux macrolides, linosamides et streptogramines B (phénotype MLSB) est un mécanisme important, notamment chez les staphylocoques. Elle se traduit par une résistance apparente à l’érythromycine mais une sensibilité à la clindamycine en l’absence d’induction.
L’antibiogramme permet de détecter ce phénotype grâce au test d’induction (ou test D). Une zone d’aplatissement du halo d’inhibition de la clindamycine à proximité du disque d’érythromycine indique une résistance inductible. Cette information est cruciale car l’utilisation de clindamycine chez ces souches peut conduire à un échec thérapeutique par induction de la résistance.
Résistance à la méticilline (SARM)
La résistance à la méticilline chez Staphylococcus aureus (SARM) est un enjeu majeur en infectiologie. Elle est due à la production d’une protéine de liaison aux pénicillines modifiée (PLP2a), codée par le gène mecA . L’antibiogramme permet de détecter les SARM par leur résistance à l’oxacilline ou à la céfoxitine, utilisées comme marqueurs.
La détection des SARM est cruciale car elle implique une résistance à toutes les β-lactamines, à l’exception des nouvelles céphalosporines anti-SARM. Elle oriente vers l’utilisation d’autres classes d’antibiotiques comme les glycopeptides ou les oxazolidinones.
Applications cliniques de l’antibiogramme
L’antibiogramme est un outil central dans la prise en charge des infections bactériennes. Ses applications cliniques sont multiples et essentielles pour une antibiothérapie raisonnée.
Choix de l’antibiothérapie empirique
Bien que l’antibiogramme nécessite généralement 48 à 72 heures, il influence indirectement le choix de l’antibiothérapie empirique. En effet, la connaissance des profils de résistance locaux, issus de l’analyse des antibiogrammes antérieurs, guide les recommandations pour le traitement probabiliste des infections courantes.
Par exemple, le taux local de SARM ou d’entérobactéries productrices de BLSE orientera le choix de l’antibiothérapie empirique dans les infections sévères. Cette approche permet d’optimiser les chances de succès du traitement initial tout en préservant l’efficacité des antibiotiques de dernier recours.
Ajustement du traitement selon les résultats
L’antibiogramme permet d’affiner l’antibiothérapie initialement prescrite de manière empirique. Dès réception des résultats, le clinicien peut ajuster le traitement pour cibler spécifiquement la bactérie identifiée. Cette désescalade thérapeutique consiste souvent à passer d’un antibiotique à large spectre à une molécule plus ciblée, réduisant ainsi le risque de sélection de résistances.
Par exemple, une infection urinaire initialement traitée par céftriaxone pourra être réorientée vers de l’amoxicilline si l’antibiogramme révèle une sensibilité à cette molécule. Ce changement permet non seulement d’optimiser l’efficacité du traitement, mais aussi de préserver l’écologie bactérienne du patient en limitant l’impact sur son microbiote.
Détection des résistances émergentes
L’antibiogramme joue un rôle crucial dans la détection précoce des résistances émergentes. Il permet d’identifier rapidement des profils de résistance inhabituels ou des mécanismes de résistance nouveaux. Cette vigilance est essentielle pour adapter les stratégies thérapeutiques et mettre en place des mesures de contrôle épidémiologique.
La détection de souches productrices de carbapénémases ou de Staphylococcus aureus résistant à la vancomycine (VRSA) sont des exemples de résistances critiques dont l’identification rapide est primordiale. L’antibiogramme constitue souvent la première alerte, déclenchant des investigations complémentaires et des mesures de prévention de la dissémination.
Surveillance épidémiologique locale et nationale
Les données issues des antibiogrammes alimentent les systèmes de surveillance épidémiologique de la résistance aux antibiotiques. À l’échelle locale, elles permettent d’établir des cartographies de résistance spécifiques à chaque établissement de santé, guidant ainsi les protocoles d’antibiothérapie empirique.
Au niveau national, ces données sont agrégées pour suivre l’évolution des résistances et adapter les recommandations thérapeutiques. Des réseaux comme l’ONERBA (Observatoire National de l’Épidémiologie de la Résistance Bactérienne aux Antibiotiques) en France jouent un rôle clé dans cette surveillance. Comment ces données influencent-elles concrètement les pratiques cliniques au quotidien ?
L’antibiogramme est bien plus qu’un simple outil de diagnostic : c’est un pilier de la lutte contre l’antibiorésistance, permettant d’orienter les politiques de santé publique et d’optimiser l’usage des antibiotiques à l’échelle individuelle et collective.
Limites et perspectives de l’antibiogramme
Malgré son importance indéniable, l’antibiogramme présente certaines limites qu’il est important de connaître pour une interprétation éclairée des résultats. De nouvelles approches sont en développement pour compléter ou améliorer cette technique essentielle.
Discordances in vitro/in vivo
L’une des principales limites de l’antibiogramme réside dans les potentielles discordances entre les résultats obtenus in vitro et l’efficacité réelle du traitement in vivo. Ces différences peuvent s’expliquer par plusieurs facteurs :
- Les conditions de croissance en laboratoire ne reflètent pas toujours l’environnement complexe du site infectieux.
- Certains mécanismes de résistance peuvent s’exprimer différemment in vivo, notamment sous la pression antibiotique.
- La pharmacocinétique et la pharmacodynamique des antibiotiques peuvent varier selon les patients et les sites d’infection.
Par exemple, une bactérie peut apparaître sensible à un antibiotique sur l’antibiogramme, mais le traitement peut échouer si l’antibiotique ne diffuse pas suffisamment au site de l’infection. C’est notamment le cas pour certaines infections ostéo-articulaires ou du système nerveux central.
Détection des biofilms bactériens
L’antibiogramme classique ne permet pas d’évaluer l’efficacité des antibiotiques sur les bactéries organisées en biofilm. Or, ces structures jouent un rôle majeur dans la persistance de nombreuses infections chroniques, comme les infections sur matériel prothétique ou les infections pulmonaires chez les patients atteints de mucoviscidose.
Des techniques spécifiques, comme le Calgary Biofilm Device, sont en développement pour tester la sensibilité des biofilms aux antibiotiques. Ces approches pourraient à l’avenir compléter l’antibiogramme classique pour une meilleure prise en charge des infections liées aux biofilms. Quelles autres innovations pourraient révolutionner notre approche de l’antibiogramme dans les années à venir ?
Antibiogrammes rapides par spectrométrie de masse
La spectrométrie de masse MALDI-TOF, déjà largement utilisée pour l’identification bactérienne, ouvre de nouvelles perspectives pour la réalisation d’antibiogrammes rapides. Cette technique permet de détecter en quelques heures les modifications du profil protéique bactérien induites par l’exposition aux antibiotiques.
L’antibiogramme par MALDI-TOF pourrait réduire considérablement le délai d’obtention des résultats, passant de 18-24h à 2-4h. Cette rapidité serait particulièrement précieuse dans les situations d’urgence, comme le choc septique, où chaque heure compte pour initier un traitement efficace.
Intégration avec les techniques de biologie moléculaire
L’essor des techniques de biologie moléculaire, notamment le séquençage à haut débit, ouvre la voie à une approche plus globale de la résistance aux antibiotiques. Ces méthodes permettent de détecter directement les gènes de résistance présents dans le génome bactérien, offrant une vision plus complète du potentiel de résistance.
L’intégration des données génomiques avec les résultats de l’antibiogramme phénotypique pourrait améliorer la prédiction de l’efficacité des antibiotiques. Cette approche, parfois qualifiée d’antibiogramme génomique, permettrait notamment de mieux appréhender les résistances inductibles ou les mécanismes complexes impliquant plusieurs gènes.
L’avenir de l’antibiogramme réside probablement dans une approche multimodale, combinant les techniques phénotypiques classiques avec les nouvelles technologies moléculaires et protéomiques. Cette évolution permettra une caractérisation plus fine et plus rapide des résistances bactériennes, pour une antibiothérapie toujours plus précise et personnalisée.
En conclusion, l’antibiogramme reste un outil incontournable dans la lutte contre les infections bactériennes et l’antibiorésistance. Ses applications cliniques, de la guidance thérapeutique à la surveillance épidémiologique, en font un pilier de l’infectiologie moderne. Malgré certaines limites, les perspectives d’évolution de cette technique laissent entrevoir des progrès significatifs dans notre capacité à combattre efficacement les infections bactériennes, tout en préservant l’efficacité des antibiotiques pour les générations futures.